Die Idee
Wir – ein kleines Team aus dem Institut für Technische Mikrobiologie – haben uns die Frage gestellt, wie wir wissenschaftliche Inhalte den Studierenden der Bioingenieurswissenschaften an der Technischen Universität Hamburg besser und vor allem praxisnaher vermitteln können. Um die Praxisnähe umzusetzen, haben wir uns für den Einsatz digitaler Medien entschieden und somit war die Idee des „Virtuellen Biologie-Labors“ (ViBiL) geboren.
Neben Studierenden sollte das Simulationsspiel ViBiL ebenfalls für Lehrende, SchülerInnen und Biologie-Interessierte zugänglich sein. Bevor das Simulationsspiel jedoch zur Verfügung stand, durchlief es einen langen Entwicklungsprozess.
Die ersten Schritte
Gestartet sind wir mit der Idee, dass in einer virtuellen Umgebung ein bestimmtes Experiment, wie man es zum Beispiel in universitären Praktika oder auch im Schulunterricht findet, von den AnwenderInnen durchgeführt werden kann. Dabei stehen verschiedene Lösungen, Geräte, Enzyme, etc. zur Verfügung, die mittels eines Drag & Drop-Prinzips in der richtigen Reihenfolge aneinandergereiht werden sollten (Bild 1). Allerdings wirkte die Darstellung sowie die Gestaltung sehr minimalistisch und die Bedienoberfläche war wenig ansprechend.
Fachbereiche der Beuth-Hochschule in Berlin unter Leitung von Prof. Felix Gers und Prof. Steffen Prowe haben bereits im Jahr 2013 einen ihrer Praktikumsversuche „Herstellung von Mozzarella“ (Bild 2) als ein Simulationsspiel entwickeln lassen. Dieser Versuch wird auch im „echten“ Praktikum durchgeführt und so konnte die Simulation den Studierenden einen ersten Eindruck verschaffen. Der große Vorteil der virtuellen Darstellung: Nichts kann kaputt gehen und Fehler sind schnell verziehen. Im Notfall wird das Experiment einfach neu gestartet.
Bild 1: Erstes Gedankenexperiment des Virtuellen Biologie-Labors.
Bild 2: Das virtuelle Laborexperiment “Mozzarella-Herstellung”.
Mit Hilfe des Know-Hows und der Unterstützung der Beuth-Hochschule in Berlin sowie deren vorhandene virtuelle Laborumgebung konnten wir im Rahmen der HOOU zwei weitere virtuelle Experimente mit dem Produktionsstudio „Villa Hirschberg Online GmbH“ realisieren.
Um verschiedene Ideen und Möglichkeiten der Umsetzung zu besprechen, habe ich mich persönlich mit Prof. Prowe und Herrn Reimer (Software-Entwickler) in Berlin getroffen. Vor allem aber haben wir die Zeit genutzt, um Herrn Reimer die Laborarbeit so nahe wie möglich zu bringen. Dafür haben wir uns in den biologischen Laboren des Fachbereiches von Prof. Prowe getroffen. Nach der Vorstellung der Herangehensweise und des Ablaufs der jeweiligen Experimente hatte Herr Reimer außerdem die Gelegenheit selbst im biologischen Labor zu arbeiten, um sich einige Handgriffe und Abfolgen anzueignen. Genauso wie in der Schauspielerei, in der man sich in verschiedene Rollen hineinversetzen muss, findet sich auch ein Software-Entwickler in unterschiedlichsten Themenbereichen wieder, die er erst einmal verstehen und nachvollziehen muss. Die Basis war durch das Treffen und die Laboreinführung gelegt.
Als es anschließend zur schriftlichen Beschreibung der jeweiligen Experimente kam, für die Vorkenntnisse notwendig sind, war ich nun selbst an der Reihe die Ausführung zu übernehmen. Die Herausforderung hierbei lag nicht in der Durchführung an sich, sondern darin meine angeeignete Routine der einzelnen Experimente abzulegen. Mein Ziel war es, die wissenschaftlichen Informationen verständlich und einfach für einen Fachfremden in einer geordneten Reihenfolge darzustellen, um somit die jeweiligen experimentellen Abläufe für die Spieleentwicklung zu vereinfachen.
Die Entwicklung
Neben der Entwicklung des eigentlichen Spiels, also der virtuellen Welt durch Code-Zeilen, Assets, etc., lag die Herausforderung darin einen einwandfreien und logischen Versuchsaufbau für den Entwickler festzuhalten. Wie bereits erwähnt, habe ich die Versuche „Polymerase-Kettenreaktion“ und „Transformation von E. coli-Zellen“ bereits unzählige Male im Labor durchgeführt, doch diese Aufgaben mussten für die logische Abfolgen Schritt für Schritt im Detail durchgegangen werden.
Da Herr Reimer in Berlin ansässig ist, haben wir hauptsächlich per Videotelefonie kommuniziert. Während wir die Abläufe durchgegangen sind kamen selbstverständlich Fragen von der Entwicklerseite auf. Fragen, mit denen ich nicht gerechnet habe, da mir die Vorgänge durch meine tägliche Arbeit quasi schon in Fleisch und Blut übergegangen sind. So kam es dazu, dass von beiden Seiten aus, ein stetiger lehrreicher Austausch stattfand und nach einer Vielzahl von immer weiter optimierten Versionen des Versuchsablaufs schließlich der finale Versuch in der Programmier-Logik feststand.
Das war noch nicht alles: Neben der schriftlichen Darstellung der Experimente mussten weitere Details der spielerischen Umsetzung geplant werden. Zum einen war die richtige Position der Utensilien und vor allem deren virtuellen Abbildung ein wichtiger Aspekt. Zum anderen spielerische Elemente, wie das Drag & Drop-Prinzip und Hilfestellungs-Optionen. Des Weiteren wurden einige Elemente eingebaut, die in der wirklichen Laborarbeit unabdingbar sind. So zum Beispiel die Verwendung von Labor-Handschuhen bei bestimmten Aufgaben im Labor (Bild 3).
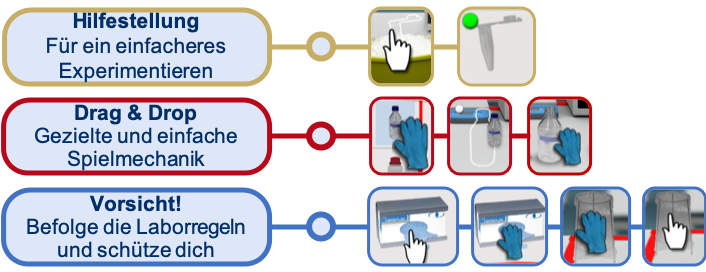
Bild 3: Mechanismen des Virtuellen Biologie-Labors. 1) Für EinsteigerInnen gibt es eine Hilfestellung, die anzeigt, welcher Gegenstand zu verwenden ist. 2) Die Mechanik des “Drag & Drop”-Prinzips ermöglicht eine einfache und gezielte Steuerung. 3) Im Labor wird Schutzkleidung getragen, deshalb ist es wichtig bei gewissen Experimenten Laborhandschuhe anzuziehen.
Ebenfalls habe ich zu jedem Versuch eine allgemeine Information für den/die AnwenderIn geschrieben, um auch Interessierte, die sich bisher nur oberflächlich mit der Thematik auseinandergesetzt haben, den Versuch so verständlich wie möglich zu beschreiben. Um den Kontext der einzelnen Versuche besser verstehen zu können, bauen diese aufeinander auf. Um dies zu verdeutlichen, wurde eine „kleine Geschichte“ – die Klonierung des grünfluoreszierenden Proteins – entwickelt, die den Experimentator anhand von die verschiedenen Laborszenen führt.
Auch bei der Entwicklung dieser ganzen Materialien gab es einen stetigen Austausch zwischen dem Entwickler, wissenschaftlichen Mitarbeitern der Beuth-Hochschule in Berlin und der Technischen Universität Hamburg. Dieser Input, sowie die Unterstützung der Universität Koblenz, führte dazu, dass sowohl die Versuchsabläufe als auch die Texte immer wieder korrigiert und optimiert wurden.
Nachdem die Inhalte feststanden, ging es zum Abschluss um die Optimierung der „Usability“, also die Bedien- und Nutzerfreundlichkeit. Dafür wurde das Spiel an den erwähnten Universitäten mit Studierenden getestet.
Die Anwendung
Zwar bin ich mit Spielekonsolen und Videospielen aufgewachsen, jedoch habe ich bis zur Entwicklung des ViBiLs mit Spieleentwicklung und -design noch keinen Kontakt gehabt. Somit war es sehr interessant zu sehen, wie die ersten AnwenderInnen die beiden Versuche Polymerase-Kettenreaktion (Bild 4) und Transformation von E. coli-Zellen (Bild 5) spielten bzw. auf welche Herausforderungen sie stießen.
Mit Hilfe dieser Tests wollte ich vor allem erfahren, welche Anregungen oder Verbesserungen die Test-AnwenderInnen für die Abläufe, die Versuche und auch die „Usability“ hatten. Dadurch konnten viele kleine „Bugs“, also Fehler, behoben werden. Einige hilfreiche Vorschläge konnten umgesetzt werden, andere wiederum waren zu komplex, als dass eine Integration ins Spiel möglich gewesen wäre. Nichts desto trotz fanden alle AnwenderInnen Gefallen an dem Spiel – genau das, was wir erreichen wollten.
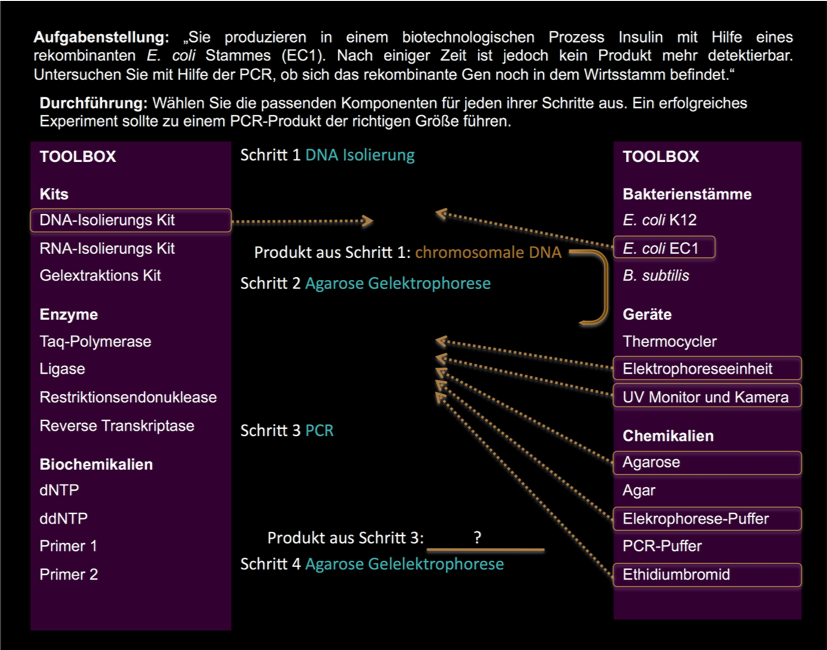
Bild 4: Das virtuelle Laborexperiment “Polymerase-Kettenreaktion (PCR)”: Das Laborexperiment “Polymerase-Kettenreaktion (PCR)”: In diesem Versuch wird neben der PCR auch eine Agarose-Gelelektrophorese durchgeführt. Da hierbei mit einer gefährlichen Chemikalie gearbeitet wird, ist es wichtig den korrekten Schutz zu gewährleisten. Deshalb werden Kittel, Laborbrille und vor allem Handschuhe verwendet.
Bild 5: Das virtuelle Laborexperiment “Transformation von E. coli-Zellen”. In diesem Versuch geht es vor allem um eine sterile Arbeitsweise. Mit dem Bunsenbrenner können sowohl Gegenstände sterilisiert werden als auch teilweise die nähere Umgebung.
Da wir an der Technischen Universität in Hamburg eben diese Versuche auch in unserem Praktikum durchführen, war es schön zu sehen, dass die Gruppen, die das Spiel vor dem eigentlichen „praktischen“ Versuch gespielt hatten, deutlich sicherer im Labor waren. Außerdem konnten sie deutlich weiterdenken und stellten Fragen, die über die eigentliche Versuchsdurchführung hinaus gingen. Dieser positive Effekt zeigt uns Möglichkeiten auf, um neben Versuchs-Praktika auch Vorlesungen anschaulicher und praxisnaher zu gestalten.
Das ViBiL könnte beispielsweise anstelle eines Kolloquiums oder schriftlichen Tests in Kombination mit weiteren Fragen als Eingangsprüfung verwendet werden. Dadurch würde der Aufwand sowohl für Lehrende deutlich reduzieren. Viele weitere Einsatzbereiche des ViBiLs sind möglich, jedoch nur, wenn das Spiel nicht lizensiert und/oder kostenfrei zugänglich ist. Das führte zur nächsten Entwicklungsphase des Spiels.
Der Open-Source Gedanke
Das ViBiL war zu Beginn nur als Desktop-Spiel verfügbar. Das bedeutete, dass es Online nicht frei zugänglich und spielbar war. Für unsere Zwecke an Universitäten oder Schulen stellte dies kein Problem dar. Jedoch war es schwierig das ViBiL auch außerhalb von Lehranstalten für InteressentInnen zur Verfügung zu stellen.
Da „MikiE – Mikroben im Einsatz“ ein Projekt der Hamburg Open Online University (HOOU) ist, wollten wir den Open-Source Gedanken für das ViBiL ebenfalls realisieren. Deshalb habe ich mich wieder mit den Entwicklern der Firma „Villa Hirschberg Online GmbH“ zusammengesetzt.
Und so startete eine weitere Entwicklungsphase in der das ViBiL zu einem spielbaren Browser-Game umprogrammiert wurde. Hierbei war ich nur indirekt beteiligt, da alle wissenschaftlichen Informationen, Abbildungen und Texte bereits durch die Desktop-Version vorhanden waren. Meine Aufgabe bestand darin neben der weiteren Leitung des Projekts auch die Testläufe zu koodinieren, um sicherzustellen, dass alle Experimente vollständig waren und keine Fehler in der Portierung zur Unity-Engine auftraten, wie z.B. fehlende Texte oder Grafiken.
Die Engine-Portierung des ViBiLs war der erste Schritt, der zweite war die Integrierung des fertigen Spiels auf die MikiE-Website. Dieser erwies sich als herausfordernder als ursprünglich gedacht und konnte durch die Unterstützung der Mitarbeiter des ITBHs (Institut für Technische Bildung und Hochschuldidaktik) erfolgreich umgesetzt werden.
Das Ergebnis dieser erfolgreichen Zusammenarbeit – das ViBiL – steht nun seit Mitte 2019 für Jede/n kostenfrei auf der MikiE-Website zur Verfügung. Eben ganz im Gedanken des Open-Source Charakters!
Die Zukunft
Das ViBiL ist nun frei zugänglich und es kann für eine Vielzahl von möglichen Einsatzbereichen verwendet werden – in universitäre Praktika, im Schulunterricht oder Zuhause. Die Umsetzung des ViBiLs ist uns erfolgreich gelungen, doch das heißt nicht, dass es keine weiteren Ideen oder Optimierungen für die Zukunft gibt.
Momentan wird an einem weiteren Experiment, der „Restriktion und Ligation von DNA“, gearbeitet, welches sich logisch in die Versuchsgeschichte eingliedert. Damit sich die AnwenderInnen noch besser in die Aufgabe hineinversetzen können, werden neue Elemente wie Videos und Fotos ergänzt, um weitere visuelle Eindrücke zu vermitteln.
Das Fazit
Die Entstehung des Virtuellen Biologie-Labors erfolgte auf einem langen, nicht geradlinig verlaufenden Weg, welcher jedoch zu stetigen Optimierungen und schließlich zur erfolgreichen Realisierung des ViBiLs führte. Ein Ende des Weges ist bisher nicht in Sicht, dafür aber weitere spannende Abzweigungen mit neuen Versuchen und Partnerschaften.
Für mich persönlich ist es eine aufregende und lehrreiche Erfahrung an diesem Projekt beteiligt zu sein. Die interdisziplinäre Zusammenarbeit mit einer Vielzahl von unterschiedlichen Menschen sowie die Vereinfachung von wissenschaftlichen Methoden und deren Beschreibung, waren sehr lehrreich für mich.
Ich bin sehr stolz auf die Entwicklungsgeschichte des ViBiLs, vor allem aber auf den Open-Source Charakter, der einzigartig in diesem Bereich ist. Ich freue mich auf die Weiterentwicklung sowie spannende und neue Herausforderungen, die dieses Projekt mit sich bringen wird.
Dieser Beitrag wurde verfasst von Dr. Philip Busch

Aktueller Podcast 42
Wir haben uns mit “42” auf den Weg gemacht und Projektinitiator_innen in Forschung und Lehre nach ihrem Werdegang befragt. Deren spannende Geschichten und Antworten hören Sie regelmäßig in unserem Podcast.

SeaPiaC
The aim of the project is to create a digital collaborative learning environment in which students of TUHH and NCKU collaborate on challenges of sustainable nature-based coastal protection in times of a changing climate.

IRIS: Individual Resorbable Intestinal Stents 2020
Ziel des Projektes ist es Menschen künftig helfen zu können, deren Heilungsaussichten und weitere Lebensqualität bislang nicht gegeben sind. Dazu sollten am Ende des Projektes Prototypen für die resorbierbaren Darm-Stents zur Verfügung stehen.

AKWAS 4.0 – Das war das Jahr 2020
AKWAS 4.0 ein Lernangebot für wissenshungrige Klimawandelbegegner mit dem Fokus Anpassung an den Klimawandel in wasserwirtschaftlichen Systemen. Reflexionen aus dem Projektjahr 2020.